nrf2 classification - 1에 이어서 데이터 중 Comment열의 3가지 값이 있다고 했는데 그 중 active열만 분포를 살펴보고 classification을 진행하였다.
data_inactive = data[data['Comment'] == 'inactive']
data_active = data[data['Comment'].str.lower() == 'active']
data_inconclusive = data[data['Comment'] == 'inconclusive']
우선 active열의 데이터에 중복값이 있는지 확인을 했다.
data_active['pIC50'].value_counts()
다음과 같이 중복되는값이 많았고 pIC50값은 같으나 smiles식이 다른 데이터들을 확인할 수 있었고 이후 active열에만 해당하는 데이터들을 csv파일로 다시 저장하였다.
# active 열만 저장
data_active.to_csv('nrf2_data_active.csv')
중복값 갯수가 상위 5개에 해당하는 데이터들을 통해 이들의 분포가 비슷한지 확인하기 위해 scatterplot을 찍어보았다.
# active내에서 scatterplot 찍기(pIC가 상위 5개)
# pIC50 값이 특정 값 중 하나인 데이터 필터링
pIC50_first_value = -4.313787100404889
pIC50_second_value = -4.363786790535726
pIC50_third_value = -4.263787512734761
pIC50_fourth_value = -4.213785953938939
pIC50_fifth_value = -4.163787309962003
# 관심 있는 pIC50 값
pIC50_values = [pIC50_first_value, pIC50_second_value, pIC50_third_value, pIC50_fourth_value, pIC50_fifth_value]
# 각 pIC50 값에 대해 scatter plot 그리기
for value in pIC50_values:
subset = data_active[data_active['pIC50'] == value]
plt.scatter(subset['Molecular Weight'], subset['AlogP'], label=f'pIC50: {value}')
plt.xlabel('Molecular Weight')
plt.ylabel('AlogP')
plt.title('Scatter Plot of Molecular Weight vs ALogP : top 5 pIC50 Values')
plt.legend()
plt.show()
이후 좀 더 세분화된 classification을 위해 inconclusive데이터를 제외한 classification과 포함한 classification 2가지를 모두 진행하였다. 정확한 classification을 위해서는 데이터의 분포가 정규분포에 가까워야 했고 두 classification의 차이를 보기 위해 나눠서 진행했다.
일단 inconclusive가 포함된 classification을 먼저 진행하였다. 데이터 분포를 보기 위해 히스토그램을 보았고 대체적으로 정규분포 그래프에 따르는 것을 확인했다.
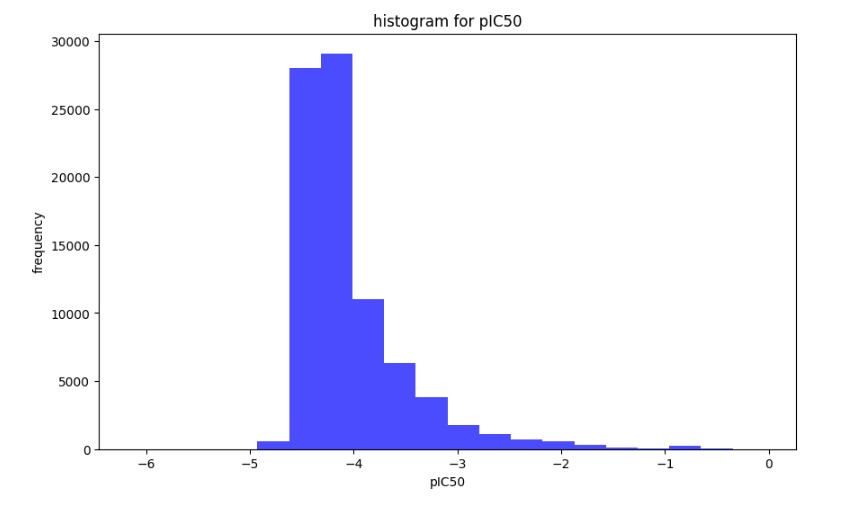
이후 0과 1로 라벨링을 하기 위해 데이터의 median값을 확인했고 그 값과 거의 유사한 -4.25기준으로 0과 1의 라벨링 데이터 갯수가 비슷하게 나타난다것을 확인하고 라벨링을 진행하였다.
nrf2_data['label'] = (nrf2_data['pIC50'] >= -4.25).astype(int)
nrf2_data['label'].value_counts()
# CSV 파일로 저장
nrf2_data.to_csv('nrf2_data_selected_label.csv', index=False)
smiles 문자열 -> fingerprint를 만들기 위해 다음과 같은 코드를 작성하였다.
# SMILES -> fingerprint
def generate_fingerprints(smiles):
mol = Chem.MolFromSmiles(smiles)
if mol is None:
return None
fingerprints = {
'fingerprint_atompair': rdMolDescriptors.GetHashedAtomPairFingerprintAsBitVect(mol),
'fingerprint_avalon': pyAvalonTools.GetAvalonFP(mol),
'fingerprint_morgan': rdMolDescriptors.GetMorganFingerprintAsBitVect(mol, radius=2),
'fingerprint_topological': Chem.RDKFingerprint(mol)
}
return {k: v.ToBitString() for k, v in fingerprints.items()}
# smiles 식 계산
for index, row in nrf2_data.iterrows():
fps = generate_fingerprints(row['Smiles'])
if fps is not None:
for fp_type, fp_value in fps.items():
nrf2_data.at[index, fp_type] = fp_value
# csv 파일 저장
nrf2_data.to_csv('nrf2_data_label_fp.csv', index=False)
그런 다음 train_test_split을 통해 train, test 데이터 셋을 분리했다.
nrf2_data = pd.read_csv('nrf2_data_label_fp.csv')
train_data, test_data = train_test_split(nrf2_data, test_size=0.2, random_state=42)
# 훈련 세트와 테스트 세트를 CSV 파일로 저장합니다.
train_data.to_csv('train_label_fp.csv', index=False)
test_data.to_csv('test_label_fp.csv', index=False)
train_data, test_data
binary classification을 진행하기 위해 다음과 같은 코드를 작성하였다.
성능 비교를 위해 RandomForestClassifier와 KNeighborsClassifier 두 가지로 나눠서 진행하였다.
결과는 다음과 같이 나왔으며 jupyterlab에서 실행하기엔 오랜 시간이 걸릴거같아 linux서버에서 분석을 진행하였다.
# fingerprint 변환 함수 : binary -> list
def convert_fingerprint(data, fingerprint_type):
return np.array([list(map(int, list(fp))) for fp in data[fingerprint_type]])
# fingerprint
fingerprint_types = ['fingerprint_atompair', 'fingerprint_avalon', 'fingerprint_morgan', 'fingerprint_topological']
# label
y_train = train_data['label'].values
y_test = test_data['label'].values
# RandomForestClassifier - HyperParameter
params = {
'n_estimators': [100, 200, 300],
'max_depth': [10, 20, 30],
'min_samples_split': [2, 5],
'min_samples_leaf': [1, 2]
}
# 최적화 및 모델 평가
for fp_type in fingerprint_types:
X_train = convert_fingerprint(train_data, fp_type)
X_test = convert_fingerprint(test_data, fp_type)
# RandomSearchCV
rf_classifier = RandomForestClassifier(random_state=42)
random_search_cv = RandomizedSearchCV(rf_classifier, param_distributions=params, n_iter=10, cv=3, random_state=42)
random_search_cv.fit(X_train, y_train)
# best model
best_rf_classifier = random_search_cv.best_estimator_
y_pred = best_rf_classifier.predict(X_test)
# 성능 평가
accuracy = accuracy_score(y_test, y_pred)
report = classification_report(y_test, y_pred)
fpr, tpr, thresholds = roc_curve(y_test, best_rf_classifier.predict_proba(X_test)[:, 1])
roc_auc = auc(fpr, tpr)
# TP, FP, FN, TP 정의(specificity 없어서 직접 계산)
conf_matrix = confusion_matrix(y_test, y_pred)
tn, fp, fn, tp = conf_matrix.ravel()
# specificity
specificity = tn / (tn + fp)
# 결과 출력
print(f"\n--- Fingerprint Type: {fp_type} ---")
print("Best Parameters:", random_search_cv.best_params_)
print("Accuracy:", accuracy)
print("Specificity:", specificity)
print("Classification Report:")
print(report)
# ROC Curve 플로팅
plt.figure()
plt.plot(fpr, tpr, color='darkorange', lw=2, label=f'ROC curve (area = {roc_auc:.2f})')
plt.plot([0, 1], [0, 1], color='navy', lw=2, linestyle='--')
plt.xlim([0.0, 1.0])
plt.ylim([0.0, 1.05])
plt.xlabel('False Positive Rate')
plt.ylabel('True Positive Rate')
plt.title(f'ROC Curve - {fp_type}')
plt.legend(loc="lower right")
plt.show()

다음과 같은 결과가 나왔으며 대체로 지표가 0.6~0.61에서 나온것을 확인할 수 있었고 정확도가 다소 떨어진다는 점을 확인하였다. 따라서 이 데이터를 이용해 regression이나 다른 회귀 분석은 조금 어려운 것으로 판단되었다.
# fingerprint 변환 함수 정의
def convert_fingerprint(data, fingerprint_type):
return np.array([list(map(int, list(fp))) for fp in data[fingerprint_type]])
# 모든 지문 유형에 대한 특징을 정의
fingerprint_types = ['fingerprint_atompair', 'fingerprint_avalon', 'fingerprint_morgan', 'fingerprint_topological']
# 훈련 데이터셋과 테스트 데이터셋에서 라벨 추출
y_train_k = train_data['label'].values
y_test_k = test_data['label'].values
# KNN Hyperparameter
params_k = {
'n_neighbors': [3, 5, 7, 9],
'weights': ['uniform', 'distance'],
'metric': ['euclidean', 'manhattan']
}
# 각각의 화학 지문 유형에 대해 모델을 훈련하고 평가
for fp_type in fingerprint_types:
# 특징 벡터 변환
X_train_k = convert_fingerprint(train_data_k, fp_type)
X_test_k = convert_fingerprint(test_data_k, fp_type)
# k-NN classifier
k_classifier = KNeighborsClassifier()
random_search_cv = RandomizedSearchCV(k_classifier, param_distributions=params_k, n_iter=10, cv=3, random_state=42)
random_search_cv.fit(X_train_k, y_train_k)
# best model
best_k_classifier = random_search_cv.best_estimator_
y_pred_k = best_k_classifier.predict(X_test_k)
# 정확도 및 분류 보고서 계산
accuracy_k = accuracy_score(y_test_k, y_pred_k)
report_k = classification_report(y_test_k, y_pred_k)
fpr_k, tpr_k, thresholds_k = roc_curve(y_test_ㅏ, best_k_classifier.predict_proba(X_test_k)[:, 1])
roc_auc = auc(fpr_k, tpr_k)
# TP, FP, FN, TP 정의(specificity 없어서 직접 계산)
conf_matrix_k = confusion_matrix(y_test_k, y_pred_k)
tn_k, fp_k, fn_k, tp_k = conf_matrix_k.ravel()
# specificity
specificity = tn_k / (tn_k + fp_k)
# 결과 출력
print(f"\n--- Fingerprint Type: {fp_type} ---")
print("Accuracy:", accuracy_k)
print("Specificity:", specificity)
print("Classification Report:")
print(report_k)
'2024 동계 UST 인턴' 카테고리의 다른 글
| [UST] 분자 구조 예측 모델 만들기 - 2 (0) | 2024.02.02 |
|---|---|
| [UST] 분자 구조 예측 모델 만들기 - 1 (0) | 2024.02.02 |
| [UST] nrf2 classification - 3 (0) | 2024.01.29 |
| [UST] nrf2 classification - 1 (0) | 2024.01.23 |
| 안전성평가연구소에서 인턴십 시작 (0) | 2024.01.12 |